Genómica Funcional
Nuestra propuesta científica pretende esclarecer el papel de los pequeños ARN reguladores en la biología del cáncer humano. Además, trabajamos en estrecha colaboración con el Hospital Universitario y el Programa Nacional de Cáncer, brindando apoyo tecnológico y experimental para la investigación en Oncología Clínica y el desarrollo de nuevos biomarcadores diagnósticos en cáncer.
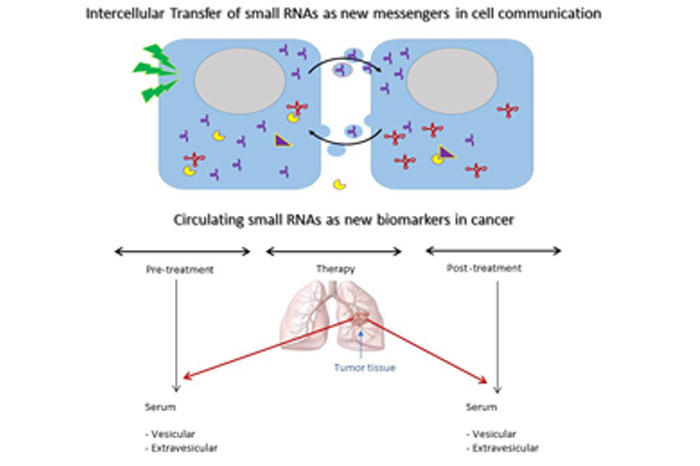
En los últimos años, nuestro foco principal de investigación se ha centrado en el estudio de una nueva clase de moléculas denominada genéricamente como pequeños ARNs no codificantes. Nuestros trabajos han demostrado que la fragmentación de estos ARNs genera moléculas capaces de regular las vías de supervivencia celular al estrés y proliferativas. Nos focalizamos principalmente en los fragmentos derivados de ARNs de transferencia e Y-RNAs, y su secreción por parte de células normales y tumorales. Estudiamos cómo otras células son capaces de captar y sensar estos ARNs liberados al medio extracelular, representando así un nuevo mecanismo de comunicación entre células. Esta y otras líneas de trabajo de la unidad están orientadas a identificar nuevas vías moleculares en la iniciación y progresión del cáncer, con especial énfasis en nuevos blancos terapéuticos y biomarcadores diagnósticos.
Integrantes



Sergio Bianchi, PhD
Investigador asociado honorario
Facultad de Medicina, Udelar sbianchi@pasteur.edu.uy

Mauricio Castellano, PhD
Posdoc institucional
Facultad de Ciencias, Udelar
mcastellano@pasteur.edu.uy








Líneas de investigación
Pequeños ARN derivados de ARNt e Y-ARNs como nuevas vías moleculares en cáncer y su potencial como fuente de nuevos biomarcadores diagnósticos en cáncer.
Nuestra principal línea de investigación, está orientada al rol de nuevas clases de pequeños ARN reguladores derivados de ARNt e Y-ARN como actores centrales en las respuestas celulares proliferativas y de supervivencia al estrés y su potencial como nuevos mecanismos moleculares en la iniciación y progresión del cáncer. El foco de nuestras actividades de investigación actuales está puesto en la biología extracelular de estos ARNs reguladores, y su capacidad de funcionar como moléculas señalizadoras entre células.
Parte de nuestros trabajos han demostrado que estos pequeños ARNs son secretados activamente por las células a través de vesículas extracelulares o fracciones extra-vesiculares, siendo transferidos a otras células constituyendo un nuevo mecanismo de comunicación intercelular y transferencia de información genética. Adicionalmente, su singular estabilidad en el medio extracelular permite su detección en fluidos biológicos posicionándose como potenciales biomarcadores moleculares séricos en cáncer humano.
Actualmente, en colaboración con el Servicio de Oncología Clínica del Hospital de Clínicas y el Instituto Nacional del Cáncer estamos estudiando el valor diagnóstico de pequeños ARNs circulantes derivados de ARNtGlu, ARNtGly e Y4-ARN en pacientes portadores de cáncer de pulmón.
Actividades de Investigación y Desarrollo con centros de Salud.
Nuestro laboratorio participa actualmente en una serie de iniciativas para incorporar nuevos biomarcadores diagnósticos en oncología, así como herramientas genómicas y moleculares.
Susceptibilidad genética al cáncer de mama. En forma conjunta con el Hospital de Clínicas de la Facultad de Medicina hemos incorporado en la rutina oncológica el estudio de mutaciones de un panel de 11 genes de predisposición hereditaria al cáncer de mama y ovario. Dicho procedimiento utiliza secuenciado profundo de última generación para el análisis incluyendo los genes BRCA1 y BRCA2 entre otros.
Biomarcadores de predicción terapéutica en cáncer de pulmón. Nuestro laboratorio realiza ensayos de hibridación in situ fluorescente (FISH) para detectar traslocaciones del gen ALK, como forma de predecir sensibilidad al tratamiento con inhibidores (Crizotinib) en cáncer de pulmón.
Cursos
- Curso internacional “Deciphering regulator RNA functions by high-throughput sequencing”. 4-8 de diciembre, 2017. Organizador: Dr. Cayota. Financiación: UNU-BIOLAC, FOCEM y auspiciantes privados.
Proyectos
2016-2018 – “Implementation of genetic tests for breast cancer risk by deep sequencing of BRCA1 and BRCA2 genes in Uruguayan women”. Fondo María Viñas – ANII.
2017-2019 – “Biosensores para la detección descentralizada de exosomas y virus del Dengue. Responsable: Juan Pablo Tosar. Financia: CSIC, Universidad de la República.
2017-2019 – “tRNA-derived small RNAs as mediators of survival and growth signals”. Responsable: Alfonso Cayota. Financia: CSIC, Universidad de la República.
2009-2010 – Integrante del equipo uruguayo en el Proyecto Multicéntrico Piloto sobre el Cáncer de Mama en Latinoamérica con el Instituto Nacional del Cáncer de los EEUU y sus contrapartes de Brasil, Argentina, México, Chile y Uruguay. Participantes: Programa Nacional de Control de Cáncer – Ministerio de Salud Pública; Facultad de Medicina (Udelar), Programa de Investigación sobre Cáncer del Institut Pasteur de Montevideo.
2009-2010 – “Helicasas con cromodominio en la iniciación y progresión de procesos leucémicos”. Fondo Clemente Estable – ANII.
2009-2010 – “MicroRNA-dependent chromohelicases as a novel tumorigenic pathway in human cancer”. The Pasteur – Weizmann Joint Research Program.
2008-2010 – “Identificación de nuevos mecanismos moleculares de carcinogénesis humana mediados por micro-ARNs y proteínas remodeladoras de la cromatina”. Comisión Sectorial de Investigación Científica (CSIC).
2008-2009 – “Análisis del gen miR-181 como marcador molecular en la iniciación y progresión de la Leucemia Linfoide Crónica”. Comisión Honoraria de Lucha contra el Cáncer (CHLCC).
Publicaciones
vacio
2018
- Fromm, B; Kang, W; Rovira, C; Cayota, A; Witwer, KW; Friedländer, M and Tosar, J.P. (2018): Plant microRNAs in human sera are likely contaminants. Journal of Nutritional Biochemistry (In press).
- Fromm, B.; Tosar, J.P.; Yu, L.; Halushka, M.; Witwer, K. (2018) miR-21-5p and miR-30a-5p are identical in human and bovine, have similar isomiR distribution, and cannot be used to identify xenomiR uptake from cow milk. Journal of Nutrition. Accepted manuscript, (In press).
- Tosar, J.P., Gambaro, F., Darre, L., Pantano, S., Westhof, E. and Cayota, A. (2018) Dimerization confers increased stability to nucleases in 5′ halves from glycine and glutamic acid tRNAs. Nucleic Acids Res. gky495; doi: 10.1093/nar/gky495
- Tosar, J.P., Rovira, C. and Cayota, A. (2018) Non-coding RNA fragments account for the majority of annotated piRNAs expressed in somatic non-gonadal tissues. Communications Biology, 1, 2.
- Tosar, J.P. and Cayota, A. (2018) Detection and Analysis of Non-vesicular Extracellular RNA. Methods Mol Biol, 1740, 125-137.
- Fagúndez, P.; Brañas, G.; Cairoli, E.; Laíz, J. andTosar, J.P. (2018) An electrochemical biosensor for rapid detection of anti-dsDNA antibodies in absolute scale. Analyst, 143, 3874-3882
2017
- Tosar, J.P., Cayota, A., Eitan, E., Halushka, M.K. and Witwer, K.W. (2017) Ribonucleic artefacts: are some extracellular RNA discoveries driven by cell culture medium components? J Extracell Vesicles, 6, 1272832.
- Mateescu, B., Kowal, E.J., van Balkom, B.W., Bartel, S., Bhattacharyya, S.N., Buzas, E.I., Buck, A.H., de Candia, P., Chow, F.W., Das, S. et al. (2017) Obstacles and opportunities in the functional analysis of extracellular vesicle RNA – an ISEV position paper. J Extracell Vesicles, 6, 1286095.
2016
- Doldán, X., Fagúndez, P., Cayots, A., Laíz, J., Tosar, J.P. (2016) Electrochemical sandwich immunosensor for determination of exosomes based on surface marker-mediated signal amplification. Anal Chem, 88, 10466-10473
2015
- Tosar, J.P., Gambaro, F., Sanguinetti, J., Bonilla, B., Witwer, K.W. and Cayota, A. (2015) Assessment of small RNA sorting into different extracellular fractions revealed by high-throughput sequencing of breast cell lines. Nucleic Acids Res, 43, 5601-5616.
- Cairoli, E., Danese, N., Teliz, M., Bruzzone, M.J., Ferreira, J., Rebella, M. and Cayota, A. (2015) Cumulative dose of hydroxychloroquine is associated with a decrease of resting heart rate in patients with systemic lupus erythematosus: a pilot study. Lupus, 24, 1204-1209.
2014
- Tosar, J.P., Rovira, C., Naya, H. and Cayota, A. (2014) Mining of public sequencing databases supports a non-dietary origin for putative foreign miRNAs: underestimated effects of contamination in NGS. RNA, 20, 754-757.
- Garcia-Silva, M.R., Cabrera-Cabrera, F., das Neves, R.F., Souto-Padron, T., de Souza, W. and Cayota, A. (2014) Gene expression changes induced by Trypanosoma cruzi shed microvesicles in mammalian host cells: relevance of tRNA-derived halves. Biomed Res Int, 2014, 305239.
- Garcia-Silva, M.R., das Neves, R.F., Cabrera-Cabrera, F., Sanguinetti, J., Medeiros, L.C., Robello, C., Naya, H., Fernandez-Calero, T., Souto-Padron, T., de Souza, W. et al. (2014) Extracellular vesicles shed by Trypanosoma cruzi are linked to small RNA pathways, life cycle regulation, and susceptibility to infection of mammalian cells. Parasitol Res, 113, 285-304.
- Garcia-Silva, M.R., Sanguinetti, J., Cabrera-Cabrera, F., Franzen, O. and Cayota, A. (2014) A particular set of small non-coding RNAs is bound to the distinctive Argonaute protein of Trypanosoma cruzi: insights from RNA-interference deficient organisms. Gene, 538, 379-384.