Investigadores de los Institutos Pasteur de París y Montevideo, y del Instituto Clemente Estable han logrado un avance significativo en la comprensión de cómo se multiplican y crecen un grupo de bacterias que incluye a las que causan enfermedades como la tuberculosis, la lepra y la difteria. Su investigación, publicada en la prestigiosa revista Nature Microbiology, arroja luz sobre dos nuevas proteínas fundamentales para este proceso. Este nuevo conocimiento podría allanar el camino para el desarrollo de fármacos antimicrobianos y de mejoras en procesos industriales.
El trabajo fue liderado por Rosario Durán, responsable del Unidad de Bioquímica Analítica y Proteómica (un grupo mixto formado por investigadores del Institut Pasteur de Montevideo e Instituto Clemente Estable), y por Ann Marie Wehenkel, de la Unidad de Microbiología Estructural del Institut Pasteur Paris.
Conociendo las bacterias
Para multiplicarse, las bacterias crecen hasta cierto tamaño y luego se dividen en dos células idénticas. A nivel molecular, este crecimiento y división están orquestados por dos complejos de proteínas conocidos como elongasoma y divisoma. Si bien se sabe mucho sobre estos sistemas en bacterias estudiadas como modelo, para otros grupos de bacterias la información disponible es muy escasa.
Entre ellos se encuentan los Corynebacteriales, que incluyen bacterias de importancia médica e industrial, como Mycobacterium tuberculosis (que causa tuberculosis), Mycobacterium leprae (causante de la lepra), Corynebacterium diphtheriae (que provoca difteria), y Corynebacterium glutamicum (utilizada para producir ácido glutámico para saborizantes y aditivos alimentarios).
Debido a características únicas de estas bacterias, entre ellas el crecimiento polar y su compleja pared celular, se sabe poco acerca de sus mecanismos de crecimiento y división celular. Lo que se conoce, sin embargo, es que a diferencia de las bacterias modelo, este grupo carece de muchas de las proteínas que habitualmente cumplen un rol importante en esas funciones.
Por eso, inicialmente, esta investigación tenía como objetivo descubrir las piezas faltantes en el rompecabezas, esto es, identificar y caracterizar las proteínas responsables de la división y el crecimiento celular de estas bacterias.
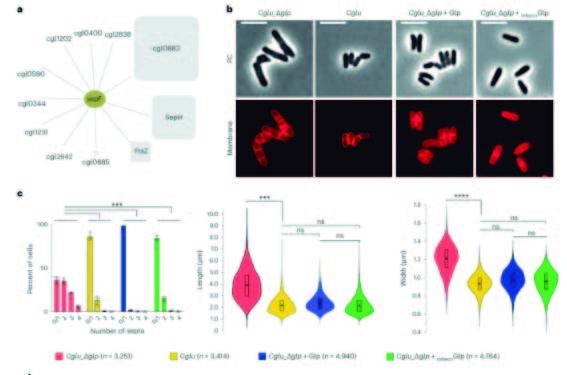
Dos nuevas protagonistas
El trabajo ha revelado por primera vez dos nuevas proteínas claves del divisoma: Glp, una molibdotransferasa, y GlpR, su receptor de membrana, que desempeñan un papel crucial en el ciclo celular bacteriano. Estas proteínas actúan como un puente, facilitando la transición del divisoma tardío en las células en división al elongasoma polar en las nuevas células hijas.
Esta investigación también revela una característica fascinante de este grupo bacteriano: la enzima Glp, originalmente involucrada en el metabolismo del molibdeno, ha experimentado una “reutilización evolutiva”, es decir, ha evolucionado con el tiempo para asumir una nueva función en un proceso biológico completamente distinto (el orquestado de una red de proteínas que gobierna la división celular).
Sorprendentemente, este fenómeno hace eco de lo que sucede en el sistema nervioso de los eucariotas superiores, donde una enzima similar llamada gefirina, también una molibdotransferasa, se ha adaptado para unirse y organizar los receptores GlyR que desempeñan un papel fundamental en la señalización sináptica. La reutilización evolutiva de las enzimas representa una de las más increíbles habilidades de la vida para responder a las condiciones cambiantes.